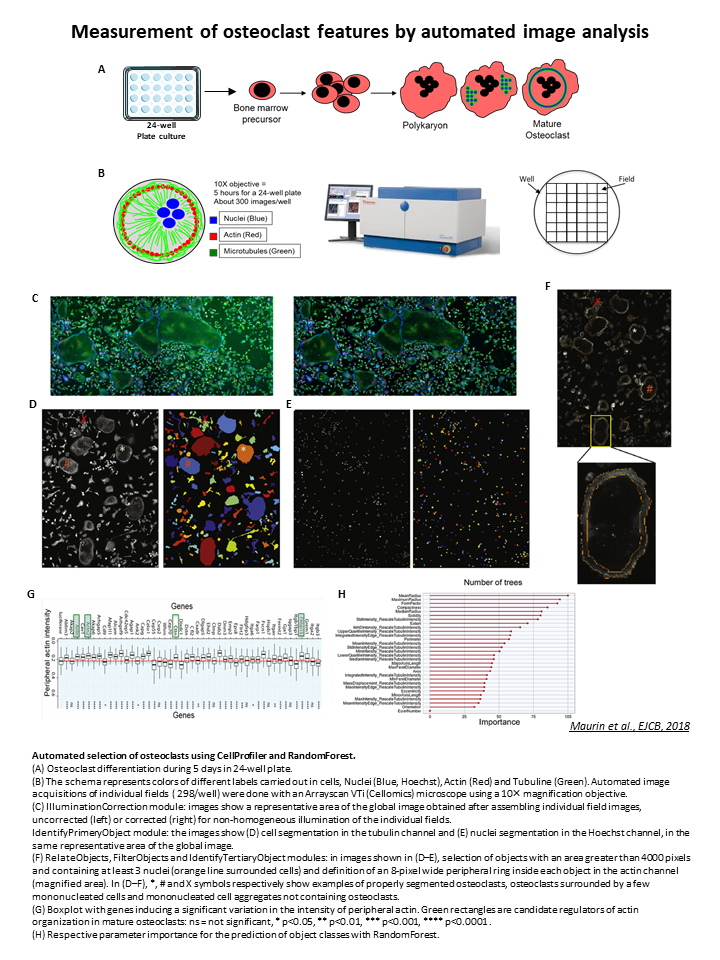
L’équipe Anne Blangy (CRBM) souhaitait réaliser un criblage pour identifier des gènes impliqués dans la différentiation cellulaire des ostéoclastes matures grâce à l’utilisation de siRNA. Pour cela, des cellules de fémur de souris sont cultivées et différenciées en ostéoclastes dans des micro-plaques 24 puits. L’ArrayScan/Cellomics a été utilisé pour les acquisitions d’images automatisées. L’objectif est de pouvoir segmenter, compter, mesurer la taille et le stade de différenciation des ostéoclastes, afin d’évaluer l’effet des siRNA testés.
Les étapes de l’analyse d’images mises en place en utilisant CellProfiler et ImageJ
- Correction d’illumination
Via CellProfiler, la correction d’illumination permet de corriger l’hétérogénéité d’illumination de chaque image individuelle sur chacun des trois canaux (Figures C et D). Vous pouvez récupérer ce pipeline sur notre github.
- Fusionner les images pour créer une mosaïque de tout le puits
Due à la taille des ostéoclastes, il est nécessaire de créer une mosaïque (globale image, Figure B) afin de conserver au maximum ces grandes structures cellulaires. Volker Baecker a donc développé une macro ImageJ afin de reconstruire cette image complète du puits. Vous pouvez récupérer cette macro sur le github de notre site ici.
- Identification des objets et quantification des paramètres
Réalisé avec CellProfiler, ce second pipeline permet l’identification/segmentation des cellules et des noyaux via les canaux Tubuline et Hoechst en utilisant le module IdentifyPrimaryObjects (Figures D et E).
Les modules MeasureObjectSizeShape, MeasureObjectIntensity et TertiaryPrimaryObjects ont permis de sélectionner les objets d’une aire supérieure à 4000 pixels², contenant au moins 3 noyaux et une couronne d’actine périphérique d’au moins 8 pixels d’épaisseur, considérés comme des ostéoclastes matures (Figures D-F).
Vous pouvez récupérer ce second pipeline sur notre github.
Ces mesures sont ensuite exportées sous R pour être analysées afin d’affiner l’identification des ostéoclastes.
Classification des objets et validation : Logiciel R
L’analyse d’image a révélé trois classes d’objets : les ostéoclastes, des ostéoclastes agrégés avec des cellules mononucléaires et des agrégats de cellules mononucléaires mal segmentées. Pour sélectionner uniquement les ostéoclastes, une classification semi-supervisée a été réalisée avec la technique dites « Random Forest ».
Pour cela, une annotation manuelle des trois classes d’objets (Figures D-F : *, #, X) dans une image mosaïque provenant d’un puits «contrôle» a été faite. La méthode Random Forest a généré un modèle avec des arbres de décisions permettant de sélectionner différents paramètres (Figure H) afin de prédire l’appartenance des objets aux trois différentes classes. Ce modèle a été appliqué sur l’ensemble du crible avec une précision de 85 % et a permis d’analyser automatiquement toutes les images du criblage.
Résultats et analyses : Logiciel R
L’effet des siRNA sur la différenciation en ostéoclastes matures a été mesuré grâce à la quantification de l’actine périphérique. Pour cela, le test statistique de Wilcoxon a été utilisé en prenant comme références les 7 siRNA contrôles du crible. Des box-plots ont été générés montrant ainsi l’effet de chaque siRNA testés par rapport aux siRNA contrôles (exemple Figure G). Cela a permis l’identification de plusieurs siRNA candidats (encadrés vert, Figure G) ayant potentiellement un rôle sur l’organisation du cytosquelette d’actine des ostéoclastes matures.